AGC Research Report 74(2024)
CystineとTyrosine添加による CHO細胞の抗体生産性の向上
Improvement of Antibody Production in CHO Cells by Addition of Cystine and Tyrosine
柴藤 祐介*
Yusuke Shibafuji
*AGC株式会社 先端基盤研究所(yusuke.shibafuji@agc.com)
Chinese hamster ovary(CHO) 細胞による抗体生産プロセス開発において、アミノ酸濃度の適切なコントロールは重要なポイントとなる。高濃度で細胞毒性を示すCysteine (Cys) や溶解度の低いTyrosine (Tyr) のようなアミノ酸を培養期間中に厳密に制御することは容易ではなく、CysやTyrの添加に関連する小胞体 (ER) ストレスや酸化ストレスといった阻害要因の細胞内メカニズムを理解したうえで、プロセスパラメーターを最適化する必要がある。
本研究では、Cysより毒性が低く、添加剤としてより一般的に用いられるCystineとTyrの添加が細胞内メカニズムに与える影響を解明するため、マルチオミクス解析 (トランスクリプトーム及びプロテオーム) を実施した。その結果、Cystineの添加は、ERストレスとER関連分解(ERAD) およびアポトーシスの促進により、生存率と生産性の低下をもたらすことが明らかとなった。一方、Tyrの添加はERストレスとアポトーシスを抑制することが確認され、この作用は、Tyrから生合成されるユビキノン (コエンザイムQ10) に起因することが示唆された。Cystine添加によってもたらされるアポトーシス促進効果を抑制するため、TyrとCystineを同時に添加したところ、GSH代謝の活性化、ERストレスと酸化ストレスの抑制、ERADの減少、クエン酸回路 (TCA cycle) の活性化などの変化が確認され、細胞の増殖、生存率が向上し、抗体生産性が向上した。 また、パスウェイ解析の結果から、DNA修復の安定化、細胞分裂の促進、酸化的リン酸化の活性化に関連する経路が、抗体産生に重要であることが示唆された。本研究で示されたマルチオミクスアプローチは、動物細胞に限らず、多様な細胞への応用も期待され、将来のモダリティも含めた多様なバイオプロセスの改善につながると考えられる。
Improvement of Antibody Production in CHO Cells by the Addition of Cystine and Tyrosine
For the monoclonal antibody(mAb) production process, controlling the concentration of amino acids is important. In particular, cysteine (Cys) and tyrosine (Tyr) are difficult to control in a culture process because of toxicity and low solubility. To achieve high productivity, the intracellular mechanisms of inhibitory events, such as endoplasmic reticulum (ER) stress and oxidative stress related to Cys and Tyr, need to be considerably elucidated. In this study, we performed multi-omics analyses (transcriptome and proteome) to compare the conditions of adding Tyr and cystine, a precursor of Cys with less cytotoxicity. The addition of cystine resulted in decreased viability and productivity because of increased ER stress, the promotion of ER-associated degradation (ERAD) and apoptosis. Conversely, the addition of Tyr suppressed ER stress and apoptosis. We thought that this effect was due to the increase in the quantity of ubiquinone (Coenzyme Q10) biosynthesized from Tyr. To inhibit the apoptosis caused by the addition of cystine, Tyr was added simultaneously with cystine. These conditions improved growth, viability, and mAb productivity because of the activation of glutathione metabolism, suppression of ER stress and oxidative stress, reduction of ERAD, and activation of the tricarboxylic acid cycle. The results of ingenuity pathway analysis suggest that pathways related to DNA repair stabilization, cell division promotion, and oxidative phosphorylation activation are important for mAb production. The multi-omics approach presented in this study will lead to improve bioprocess.
1. 緒言
近年、バイオ医薬品が注目を集めており、特に標的分子への高い特異性から抗体医薬品の開発が活発に行われている(1)。抗体生産の需要拡大に伴い、多くの企業は生産プロセスの改良に注力している。抗体生産には、一般的にCHO細胞が利用されることが多く、培養培地や培養方法の最適化により、必須栄養素の枯渇を防ぎつつ、乳酸やアンモニアの蓄積を防ぐようなプロセスデザインがなされてきた(2)。これらの研究のほとんどは、1つの要因の効果に焦点を当てており、アミノ酸等の必須成分の交互作用に関する研究はまだ限られている。また、より高い生産性を達成するためには、細胞のERストレスや酸化ストレス等の阻害要因として知られる細胞内メカニズムの解明が必要であるが、その研究はまだ不十分である。
CHO細胞による抗体生産において、重要アミノ酸濃度の厳密な制御が、高い抗体生産性を達成するために必須であることが認識されている。しかし、細胞毒性を示すCysteine (Cys) や溶解度の低いTyrosine(Tyr) のようなアミノ酸の制御は添加溶液調整の難しさや濃度コントロールがシビアであることから課題が多い(3,4)。
Cysは必須アミノ酸ではないが、細胞にとって必要不可欠であり、増殖や生存率の維持・生産性の向上に影響を与えるとの報告がこれまでに多数なされている(3,5)。しかし、pKaが8.45であるCysのチオールは生理的条件下 (pH 7.4) で比較的容易に解離し、チオラートアニオンを生じるため、細胞毒性が強く、培地中のCys濃度は低く抑えられていることが一般的である(6)。一方で、Cystineはチオール基がジスルフィド結合を形成しているため解離が生じない。また、Cystine/glutamate transporterにより容易に細胞内に輸送されるため(7)、細胞にとって安全なCys源となる。
Cysはグルタチオン(GSH) 合成の律速基質であり、Cysの枯渇は細胞内GSHの枯渇を引き起こす。GSHはトリペプチド (γ-L-グルタミル-L-システイニルグリシン) であり、活性酸素の解毒や小胞体でのジスルフィド結合形成の調節等、細胞内のERストレスや酸化ストレスの軽減作用と密接に関係している(8 - 10)。
抗体には多数のジスルフィド結合が存在するため、この結合の形成が抗体生産の律速となる。ジスルフィド結合の形成には、Protein disulfide isomerase(PDI) が必要であり、PDIは新生ポリペプチド鎖のCysと電子を授受し、酸化的フォールディングを触媒する。この過程で還元されたPDIは再酸化され、過酸化水素 (H2O2) を発生させる。発生したH2O2はGSHにより還元される。このため、GSHはH2O2の解毒に重要であり、GSHの枯渇は、酸化ストレスによるERストレスを誘導する(3,5,11)。このため、細胞内のCys濃度の低下は、GSHに関連した細胞ストレスを惹起し、抗体の生産性の減少を引き起こすと考えられているが(12)、そのメカニズムの詳細はまだ十分にわかっていない。
Tyrは比生産性 (Qp) の維持とTyr配列変異の回避に関与することがわかっており、培養時のTyr濃度の減少は、生細胞密度( VCD) の減少や、生存率の下落を引き起こす(13,14)。Tyr濃度の低下が、オートファジー性細胞死によるリソソームの酸性化と細胞溶解を生じさせる可能性が示唆されているが(4)、Tyrの枯渇によって生じる細胞ストレスや培養トレンドの変化との関連・詳細メカニズムについては、未だ明らかとなっていない。また、ベンゾキノンのキノイド核であるコエンザイムQ10は、細菌や真核微生物のシキミテート経路に由来し、哺乳類では必須アミノ酸であるTyrから合成され、酸化ストレス軽減作用があることがわかっている(15)。ただし、このようなTyr由来の生合成由来分子の細胞への影響の詳細はわかっていない。
本研究では、マルチオミクス解析により、CystineとTyr添加に伴うERストレス、酸化ストレス、TCAサイクル活性、オートファジー、アポトーシスに関連する抗体産生阻害因子を特定し、CystineとTyrの細胞への影響を明らかにすることを目的とした。
2. 実験方法
2.1. オミクス解析のためのFed-Batch培養
無血清振とう培養に適応させたDG44 CHO細胞をホスト細胞として、IgGをGOI (Gene of interest) とした抗体発現用ベクター (自社開発) を細胞にトランスフェクションした。得られたトランスフェクタントプールをシングルセルクローニングし、クローンを取得した。取得してきた抗体生産クローンを用いて、Fed-batch培養を行い、オミクス解析のためのサンプリングを実施した。
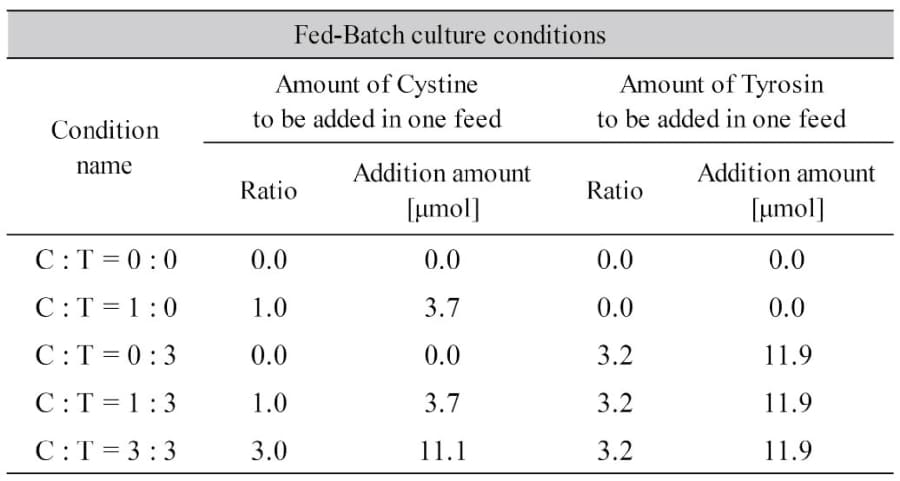
Fed-batch培養は、シェーカーインキュベーターにて、125 mLエルレンマイヤーフラスコを用いて実施した。培養は、37.0℃、125 rpm (振とう直径25mm)、5 % CO2条件で行った。独自の化学的に定義された基礎培地を使用し、開始容量 30 mLで各条件それぞれ3回の培養を実施した (Three biological replicates)。生細胞密度 0.5 x 106 cells/mLでFed-batch培養を開始し、独自の化学的に定義されたFeed培地を一定の間隔で添加した。CystineとTyrのストック溶液をそれぞれ調整し、Feed培地と同様に培養3日目から添加した。各培養条件のCystine、Tyrは標準添加量を1.0とした時の比率としてTable 1に示す量を添加した。
 参考文献(16)より改変転載
参考文献(16)より改変転載2.2. コエンザイムQ10を添加したBatch培養
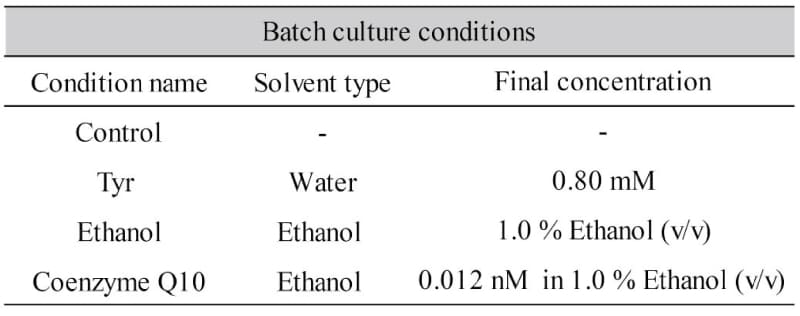
前述と同じIgG発現クローンを用いてBatch培養を行った。125 mLエルレンマイヤーフラスコを用いて、シェーカーインキュベーターにて培養を行った。CDDG44 medium (Thermo fisher : Catalog number:12610010) に、L-Glutamine、Insulin、Pluronicを添加した培地を基礎培地として使用した。培養の開始液量を25 mLとし、各培養条件でそれぞれ3回の培養を実施した(Three biological replicates)。Batch培養開始時(Day 0) に、Table 2に示した量のサプリメントを添加し、培養Day 7にサンプリングを行った。
 参考文献(16)より改変転載
参考文献(16)より改変転載2.3. 生細胞密度、生存率、生産性および 代謝物の測定
VCDと生存率の測定は、Vi-CELL XR Cell Viability Analyzer (Beckman Coulter) を用いて、トリパンブルー排除法により測定した。生産性の測定は、培養液を遠心分離 (9391 g x 1 min, 4℃) して細胞を除去し、Octet (Sartorius) を用いて測定した。また、代謝物の測定は BioProfile FLEX2(Nava biomedical) を用いて実施した。
2.4. トランスクリプトームとプロテオーム
各培養条件から、3回のサンプリングを実施した。サンプリングした培養液を遠心( 14000 g x 30 sec x 2℃) によって細胞と上清に分離し、上清を除去した。残った細胞ペレットを2 mLの氷冷したPBSで洗浄した後、再び遠心分離 (14000 g x 30 sec x 2℃)してPBSを除去し、得られた細胞ペレットサンプルを分析するまで-80℃で凍結保管した。
トランスクリプトームのシーケンシングはAzenta Life Sciences (Chelmsford, MA, United States) にて行った。細胞ペレットから全RNAを抽出し、polyA選択によってmRNAを濃縮した。mRNAをもとにライブラリーを調製し、イルミナ社NGSによってペアエンドモードでシーケンシングを行った。
RNA-seqのデータはFastp version 0.21.0 (17) によってトリミングした後、STAR version 2.7.6a (18)を用いてゲノムD N A配列 (Cricetulus griseus CHOK1GS, Ensembl release 104, accession number GCA_900186095.1) にアライメントした。アライメント結果をもとにRSEM version 1.3.3(19)を用いて転写産物のカウントを行った。発現変動遺伝子の検出はedgeR version 3.38.1(20)を用いて行った。
プロテオーム測定はThermo Scientific EASY-nLC1200(Thermo Fisher Scientific)および Thermo Scientific Orbitrap Eclipse Tribrid mass spectrometer (Thermo Fisher Scientific) を用いて行った。
LC/MSによって取得したデータはScaffold DIA version 3.1.0 (Proteome Software, Oregon, United States) を用いて解析した。スペクトルライブラリーはEnsemblから取得したアミノ酸配列(C. griseus CHOK1GS, Ensembl release 104) をもとに、Scaffold DIAとProsit(21)を用いて作成した。
パスウェイ解析はIngenuity Pathway Analysis software(IPA, QIAGEN) を用いて行った。
3. 実験結果
3.1. Cystine添加の影響 (C:T=1:0とC:T=0:0の比較)
Cystineの添加条件 (C:T=1:0) と無添加条件(C:T=0:0) の比較を行うためにFed-Batch培養およびオミクス解析を行った。Cystine添加有無の培養トレンドデータおよびオミクス解析の結果をFig. 1に示す。
先行研究とは異なり(3,5)、Cystineの添加により、培養後半に生存率および生産性が低下する傾向が確認された。C:T=1:0条件では、Day14まで生存率が維持できず、オミクス解析のための適切な細胞サンプルを取得できなかったため、C:T=1:0条件に関してはDay12までのオミクスデータをC:T=0:0条件と比較した。
Hspa5(Bip, GRP-78) およびErn1はERストレスのマーカーであることが知られている(22,23)。また、活性化転写因子3遺伝子(Atf3)はERストレスによるPERKの活性化により発現が誘導されることが知られている(24) 。これらのERストレスマーカーのmRNA量がCystineの添加により、顕著に増加していることから、Cystineの添加がERストレスの原因となっていることが示された(Fig. 1 D, E, F)。
ERAD関連因子として知られている活性化転写因子6遺伝子(Atf6)、UBX domain-containing protein 4(Ubxn4)、Suppressor/Enhancer of Lin-12-like(Sel1l) のmRNAレベルを比較した(Fig. 1 G, H, I)。Atf6は小胞体ストレス応答(UPR) に関連し、ERAD経路の構成要素であることが知られている(25)。Ubxn4はUBXD2とも呼ばれ、Ubxn4 geneにコードされるタンパク質であり、ERADを促進するER内在性膜タンパク質であることがわかっている(26)。また、Sel1lは、ERADにおいて、ミスフォールドした小胞体タンパク質のユビキチン依存性分解に関与していることが報告されている(27)。C:T=0:0条件に比べてC:T=1:0条件で、これらマーカーのmRNA量が顕著に増加していた。この結果から、Cystineの添加によって培養後半に積極的なERADが引き起こされることが明らかとなった。
また、アポトーシスに関連する因子の培養条件間の比較を行った。ERストレスによりアップレギュレーションされるアポトーシス誘導タンパク質GADD34(Ppp1r15a) は、プロテアソーム阻害による細胞死を促進することがわかっている(28)。このPpp1r15aのmRNA量が、C:T=1:0条件で顕著に上昇していることから、Cystine添加条件では、有意にアポトーシスが促進されていることが確認された(Fig. 1J)。
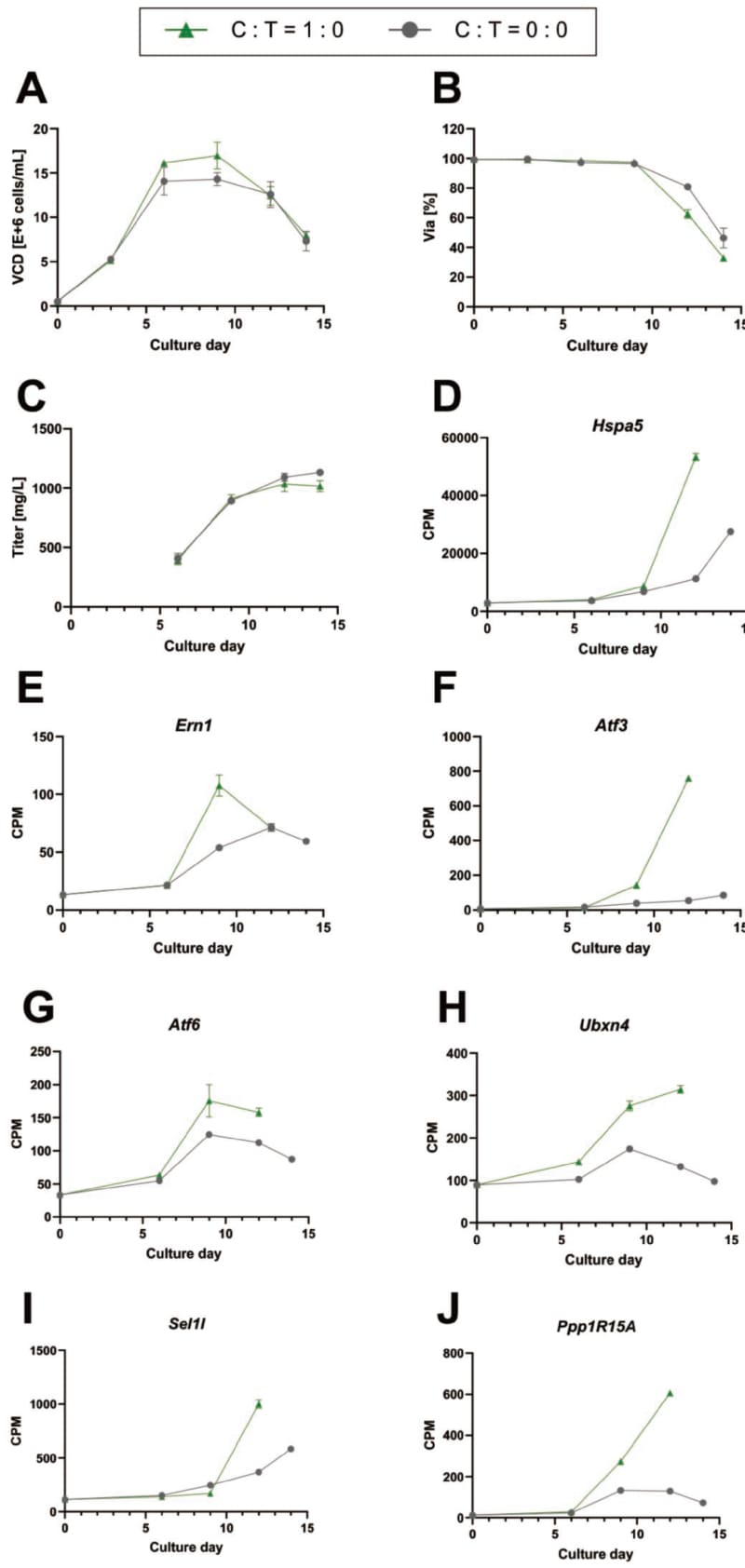 Fig. 1 The effect of Cys addition in fed-batch culture.(A) VCD profile,(B) Viability profile,(C) Titer profile,(D)–(J) Time-course plots of the associated RNA transcripts(CPM). Each value represents the mean value( N=3), and error bars represent the standard deviation. Gray(circle dots) and green(triangle dots) indicate C:T=0:0 and C:T=1:0 conditions, respectively.
Fig. 1 The effect of Cys addition in fed-batch culture.(A) VCD profile,(B) Viability profile,(C) Titer profile,(D)–(J) Time-course plots of the associated RNA transcripts(CPM). Each value represents the mean value( N=3), and error bars represent the standard deviation. Gray(circle dots) and green(triangle dots) indicate C:T=0:0 and C:T=1:0 conditions, respectively.参考文献(16)より改変転載
3.2. Tyrosine添加の影響 (C:T=0:3とC:T=0:0の比較)
先行研究では(4)、CHO細胞のFed-batch培養中、Tyrの添加によって、培養後半の生存率の減少抑制や、オートファジー性細胞死の抑制ができることが報告されている。Tyr添加が培養に与える影響について検証するため、Fed-batch培養とオミクス解析を行った (Fig. 2)。
培養結果から、先行研究と同様に、Tyrの添加は、生存率の減少を抑制できることがわかった。また、ERストレスのマーカーであるHspa5, Ern1, Atf3のmRNA量が減少していることから、Tyrの添加によってERストレスを抑制できることが確認できた(Fig. 2 D, E, F)。
先行研究で示唆されている、Tyr添加によるオートファジー抑制効果の検証として、C:T=0:0条件とC:T=0:3条件におけるP62/Sqstm1の発現量を比較した。P62/Sqstm1はLC3と直接結合することで選択的にオートファゴソームに取り込まれ、オートファジーによって効率的に分解されることがわかっている。そのため、オートファジーが阻害されると蓄積し、オートファジーが誘導されると減少する(29)。Fig. 2Gの結果から、P62/Sqstm1のmRNA発現量をTyr添加条件 (C:T=0:3) とTyr無添加条件 (C:T=0:0) 間で比較したところ、ほぼ同程度であることがわかった。この結果は、Tyr添加の有無によってオートファジーの頻度に差が無いことを示している。一方、Ppp1r15aのmRNA発現量がTyr添加条件 (C:T=0:3)で減少していることから、Tyr添加によるアポトーシスの抑制は確認された(Fig. 2H)。
Tyr添加によるアポトーシスの抑制・生存率の維持作用がどのようなメカニズムで生じているかを検証するため、Tyrから生合成されるコエンザイムQ10に着目した。コエンザイムQ10が酸化ストレスを軽減させ、アポトーシスを抑制している可能性を検証するため、Tyr無添加のBatch培養中にコエンザイムQ10を添加し、Tyr添加の条件と同様の結果が得られるかを検証した (Fig. 2 I, J)。結果、コエンザイムQ10の添加は、Tyr添加と同レベルの生産性をもたらすことが確認され、Tyr添加条件よりも生存率の低下を防げることがわかった。この結果は、Tyrから生合成されるコエンザイムQ10が培養後半の生存率の維持、具体的には、酸化ストレスの減少やアポトーシスの抑制に効果があるという仮説を支持するものと思われる。
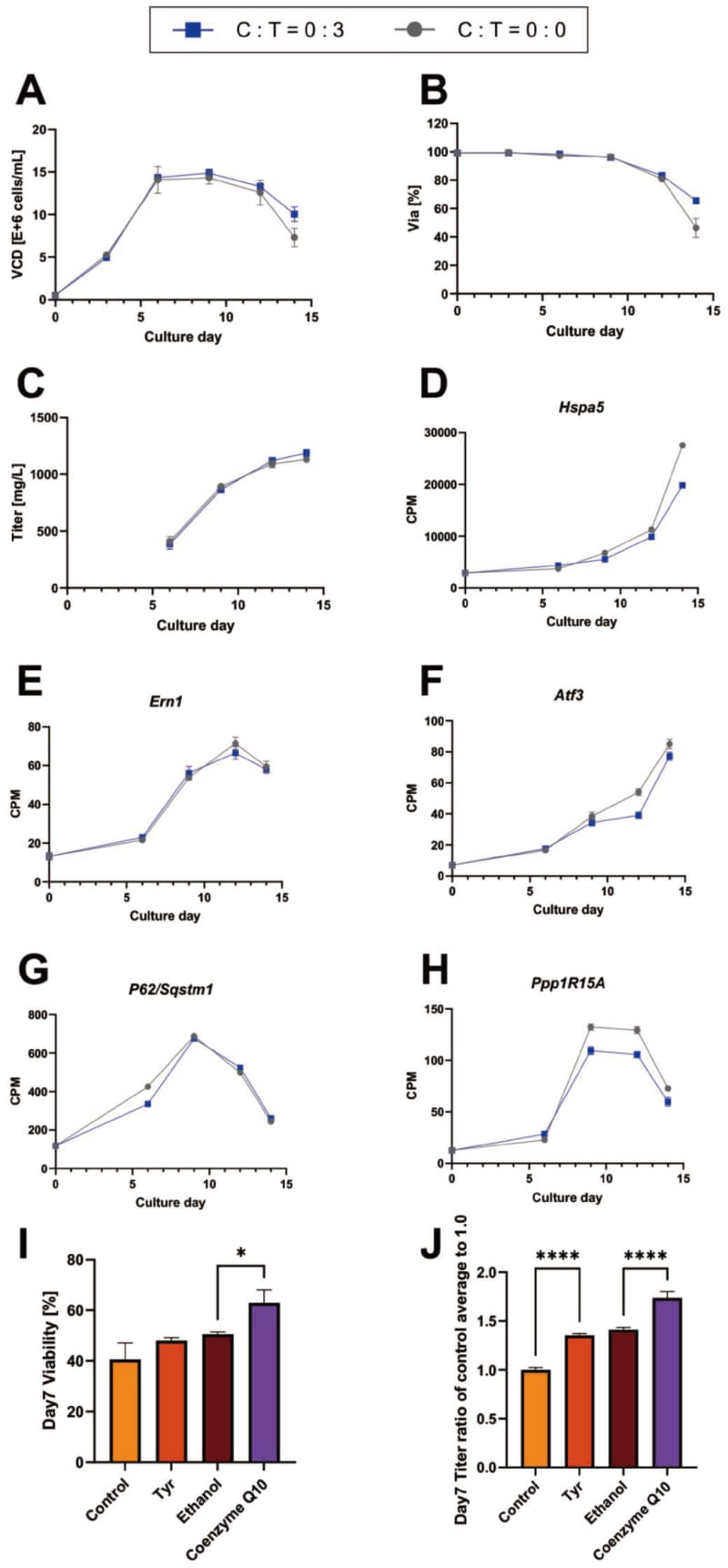 Fig. 2 The effect of Tyr addition in fed-batch culture.(A)VCD profile,(B) Viability profile,(C) Titer profile,(D)–(H) Time-course plots of the associated RNA transcripts(CPM). Each value represents the mean value(N = 3), and error bars represent the standard deviation. Gray(circle dots) and blue(square dots)indicate C:T=0:0 condition and C:T=0:3 conditions, respectively.(I) and(J) Viability and productivity data with coenzyme Q10 addition. Each value represents the mean value( N=3), and error bars represent the standard deviation(* : P value<0.0274,**** : P value <0.0001).
Fig. 2 The effect of Tyr addition in fed-batch culture.(A)VCD profile,(B) Viability profile,(C) Titer profile,(D)–(H) Time-course plots of the associated RNA transcripts(CPM). Each value represents the mean value(N = 3), and error bars represent the standard deviation. Gray(circle dots) and blue(square dots)indicate C:T=0:0 condition and C:T=0:3 conditions, respectively.(I) and(J) Viability and productivity data with coenzyme Q10 addition. Each value represents the mean value( N=3), and error bars represent the standard deviation(* : P value<0.0274,**** : P value <0.0001).参考文献(16)より改変転載
3.3. Cystine とTyrosineの同時添加の影響 (C:T=0:3、C:T=1:3、C:T=3:3の比較)
Cystine添加による還元力低下から生じるアポトーシスを抑制しつつ、十分量のCysを細胞に供給するため、Cystineと同時にTyrを添加し、Cystineの量比を変えた実験を行った。また同時に、CystineとTyrの相乗効果を明らかにするため、トランスクリプトームとプロテオーム解析を実施した(Fig. 3 - 4)。
Cystineのみ添加した条件(C:T=1:0条件, Fig. 1) とは異なり、十分なTyrを同時添加することで(C:T=1:3, C:T=3:3)、増殖能が向上し、生存率が高く維持され、生産性が向上することがわかった (Fig. 3 A, B, C)。最終的な生産性はC:T=0:3条件の生産性を基準として、C:T=1:3条件では約2倍、C:T=3:3条件では約3倍も生産性が向上した。C:T=3:3条件は、C:T=1:3条件と比べて、増殖も大幅に改善されている。これらの結果から、CHO細胞のCystineの消費にはTyrが必要であることが示唆された。
また、ERストレスマーカーであるHspa5, Ern1,Atf3の結果より (Fig. 3 D, E, F)、C:T=0:3条件と比べて、C:T=1:3条件とC:T=3:3条件で、ERストレスマーカーのmRNA量が培養後半に低い。このことから、CystineとTyrの十分量の添加は、ERストレスを抑える効果があることがわかった。
抗体はジスルフィド結合が多く、複雑な立体構造をしたタンパク質であり、その生産にはPDI等の分子シャペロンが欠かせない。Fig. 3 G, Hの結果から、PDIの発現量がCystineとTyrを必要量添加することで上昇することがわかった。この結果から、細胞内で分子シャペロンが適切にワークし、タンパク質の適切なフォールディングがなされることによって、ミスフォールディングタンパク質の滞留によるUPRを防ぎ、抗体の生産性を向上できていると考えられる。
Superoxide dismutase 1 and 2 (SOD1, SOD2)は、それぞれ細胞質およびミトコンドリアでスーパーオキシドアニオンラジカルをより害の少ないH2O2に変換する酵素であり、酸化ストレスおよび細胞毒性から細胞を保護するのに役立つことが分かっている(30,31)。SOD1, SOD2のタンパク質発現量は、C:T=0:3条件に比べて、C:T=1:3条件とC:T=3:3条件にて増加している (Fig. 3 I, J)。この結果から、C:T=1:3条件とC:T=3:3条件では、酸化ストレスに適切に対処できていることがわかった。適切なシャペロンの発現と、それに伴う酸化ストレスへの対処が豊富なCysから生じるGSHおよびSODにより達成できている可能性が考えられた。
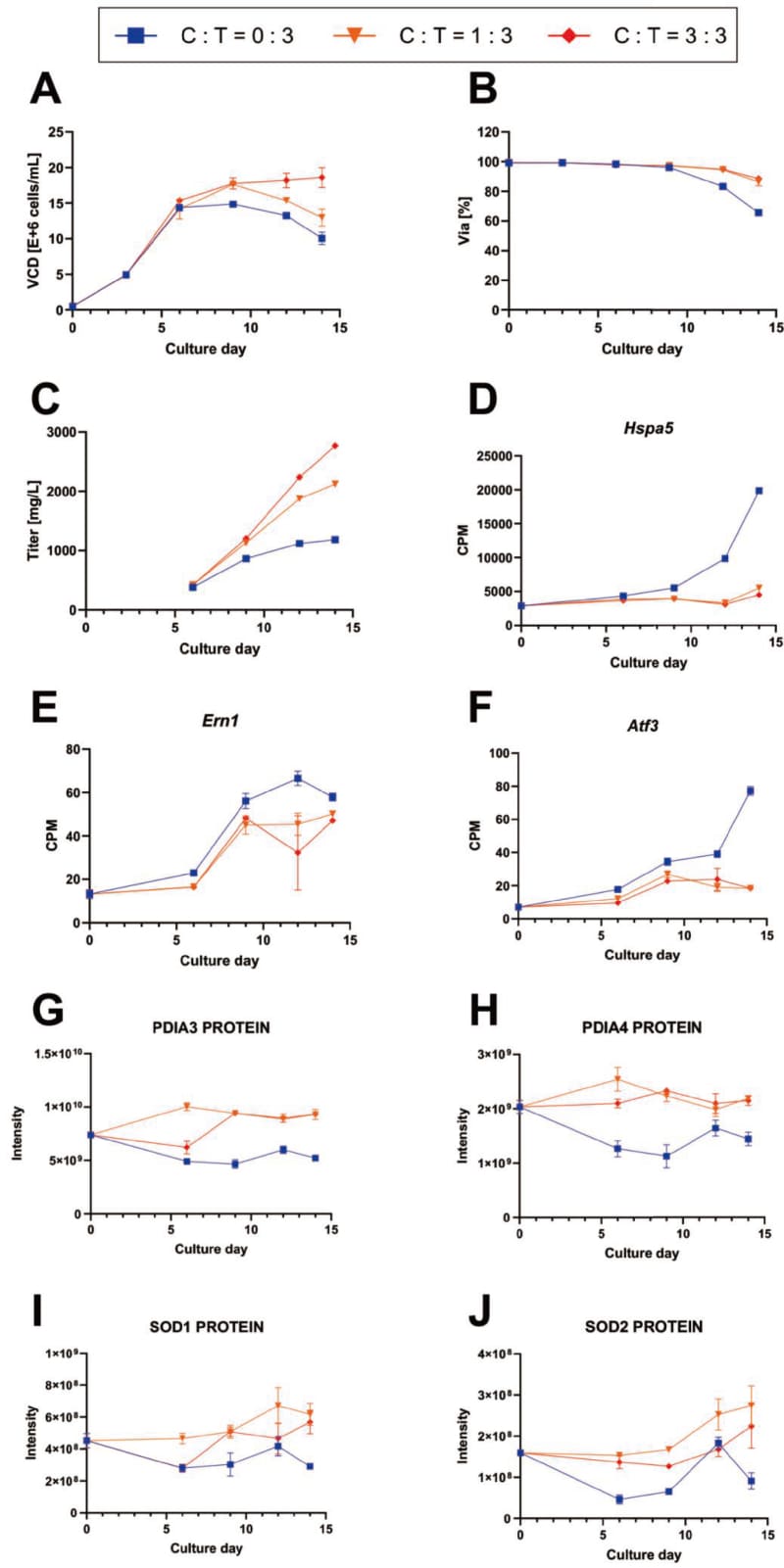 Fig. 3 The synergistic effects of cystine and Tyr in fed-batch culture.(A) VCD profile,(B) Viability profile,(C) Titer profile,(D) –(J) Time-course plots of the associated RNA transcripts(CPM) and proteins(abundance). Each value represents the mean value (N = 3), and error bars represent the standard deviation. Blue(square dots), orange(inverted triangular dots), and red(diamond dots) indicate the C:T=0:3, C:T=1:3 and C:T=3:3 conditions, respectively.
Fig. 3 The synergistic effects of cystine and Tyr in fed-batch culture.(A) VCD profile,(B) Viability profile,(C) Titer profile,(D) –(J) Time-course plots of the associated RNA transcripts(CPM) and proteins(abundance). Each value represents the mean value (N = 3), and error bars represent the standard deviation. Blue(square dots), orange(inverted triangular dots), and red(diamond dots) indicate the C:T=0:3, C:T=1:3 and C:T=3:3 conditions, respectively.参考文献(16)より改変転載
次に、GSH代謝関連因子の比較を行った(Fig. 4 A, B, C)。GSH代謝に関連する酵素として、膜外酵素γ-グルタミルトランスフェラーゼ(GGT) とグルタチオンS-トランスフェラーゼ(GST) がある。GGTは、細胞外 GSH のグルタミン酸とCysの間の γ-グルタミル結合の加水分解を触媒し、GSHの分解を促進させる酵素である(32)。GSTMは、μファミリーに分類されるGSTであり、GSHと求電子分子との結合を触媒することで、細胞内の解毒を行うことがわかっている(33)。Fig. 4の結果から、C:T=0:3条件でGgt1のmRNA発現量が増加していることがわかった。この結果から、Cysの供給がないC:T=0:3条件においては、細胞内にCysを取り込むために、GSHを積極的に分解していることが示唆された。一方で、C:T=1:3条件とC:T=3:3条件では、細胞内Cys量が十分であるため、Ggt1の発現量が抑えられ、GSHの分解が抑制されていることがわかった。また、Gstm2, Gstm7のmRNA量が、C:T=0:3条件と比較して、C:T=1:3条件とC:T=3:3条件で多くなっていることから、C:T=1:3条件とC:T=3:3条件では細胞内の解毒作用が活性化されていることがわかった。これらの結果から、C:T=1:3とC:T=3:3の条件では、分解されずに残ったGSHをH2O2の還元や細胞の解毒に利用することができ、細胞活動をより正常化および活性化することができていると考えられた。
抗体の生産性に影響を与えるERAD関連因子の比較を行うため、前出しているAtf6, Ubxn4, Sel1lのmRNA量を解析した(Fig. 4 D, E, F)。これらの因子の発現量がC:T=0:3条件で顕著に多く、Atf6とSel1lに関してはC:T=1:3条件とC:T=3:3条件の結果で見られるようにCystineの添加量を多くすることに伴って、ERAD関連因子の発現量が減少することがわかった。この結果から、TyrとCystineの十分量の添加は、ERストレス・酸化ストレスを抑制し、ERADを抑えることができることがわかった。
最後に、細胞の増殖や生存率に影響を与えるTCA回路関連因子とアポトーシス関連因子の比較解析を行った (Fig. 4 G, H, I, J)。TCA回路活性化のための重要な酵素として、コハク酸デヒドロゲナーゼ (SDH)およびアコニターゼ (ACO) がある。SDHは、クエン酸サイクル中で、コハク酸をフマル酸に酸化する機能があり、SDHA・SDHBはSDHの触媒コアを形成する2つのサブユニットである(34)。ACOはTCAサイクル中でクエン酸をイソクエン酸に変換する酵素であり、ACO2はミトコンドリアで発現することが知られている。ACOの活性は、酸化ストレスのバイオマーカーとしても良く知られており、酸化還元状態のミトコンドリア内センサーとして機能することがわかっている(35)。Fig. 4 G, H, IのSDHA・SDHBおよびACO2のタンパク質発現量のデータから、C:T=0:3条件に比べて、C:T=1:3条件とC:T=3:3条件では、SDHA・SDHBおよびACO2の培養後半での発現量が高く、培養後半までTCA回路が活性化できていることが示唆される。また、アポトーシスマーカーのPpp1r15aの結果から、C:T=0:3条件と比較して、C:T=1:3条件とC:T=3:3条件では、Ppp1r15aのmRNA量が減少傾向であることから、C:T=1:3条件とC:T=3:3条件にてアポトーシスが抑制されていることがわかった。
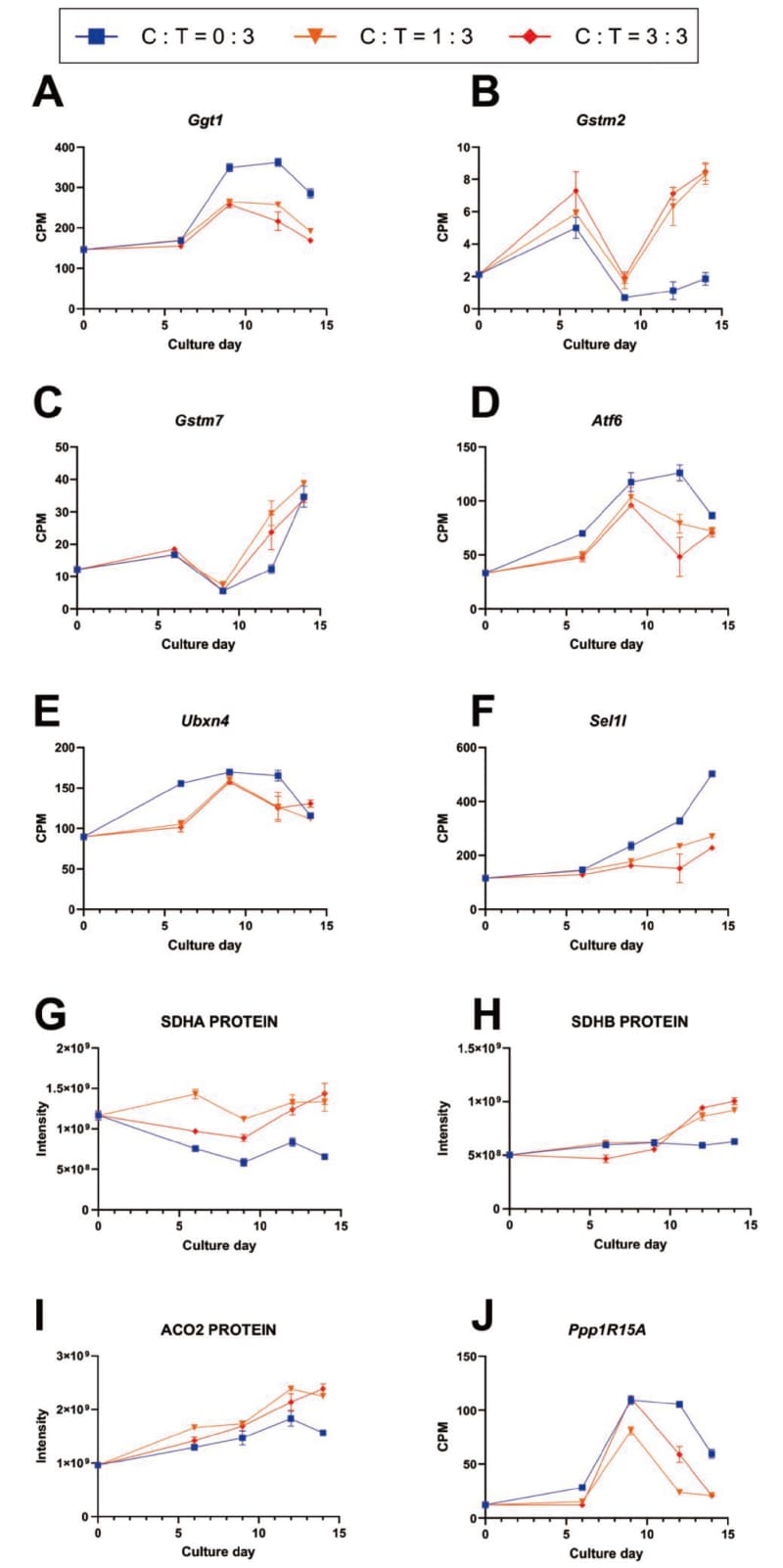 Fig. 4 The effects of cystine and Tyr.(A) –(J) Time-course plots of the associated RNA transcripts(CPM) and proteins(abundance). Each value represents the mean value(N=3), and error bars represent the standard deviation. Blue(square dots), orange(inverted triangular dots), and red(diamond dots)indicate the C:T=0:3, C:T=1:3 and C:T=3:3 conditions, respectively.
Fig. 4 The effects of cystine and Tyr.(A) –(J) Time-course plots of the associated RNA transcripts(CPM) and proteins(abundance). Each value represents the mean value(N=3), and error bars represent the standard deviation. Blue(square dots), orange(inverted triangular dots), and red(diamond dots)indicate the C:T=0:3, C:T=1:3 and C:T=3:3 conditions, respectively.参考文献(16)より改変転載
オミクス解析結果から、C:T=0:3条件と比べて、C:T=1:3条件とC:T=3:3条件では、細胞内ストレスが同程度抑制されていることがわかったが、C:T=1:3とC:T=3:3では、増殖・生産性に差がある。この理由を明らかにするため、全トランスクリプトームデータの発現変動レベルを解析した (トランスクリプトーム解析のデータからFDR < 0.05の遺伝子を抽出した)。Day6, Day9, Day12, Day14において、発現変動が見られた遺伝子の数をFig. 5にまとめた。この結果から、Day 12が最も発現変動遺伝子の数が多く、培養条件間の細胞内パスウェイの活性化度合いに最も差があることがわかった。
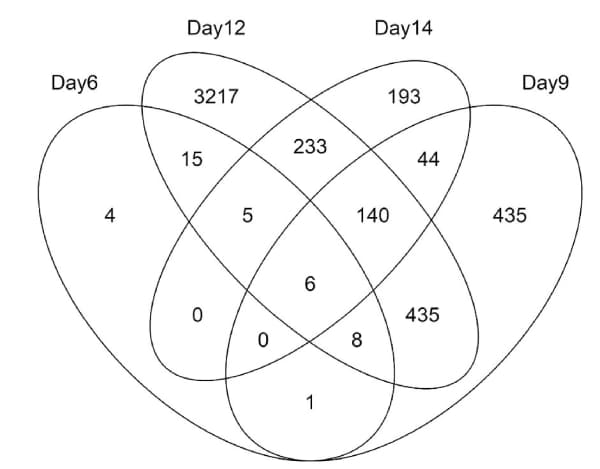 Fig. 5 Number of genes with variable mRNA expression for each culture day in the C:T=1:3 and C:T=3:3 conditions(genes with FDR < 0.05 were extracted from the transcriptome analysis data).
Fig. 5 Number of genes with variable mRNA expression for each culture day in the C:T=1:3 and C:T=3:3 conditions(genes with FDR < 0.05 were extracted from the transcriptome analysis data).次に、発現変動遺伝子数が最も多かったDay12のデータについて、C:T=1:3条件とC:T=3:3条件の間でどのようなパスウェイがアクティベートされているかを調べるため、パスウェイ解析を行った。同定された533のパスウェイの中、C:T=1:3条件に対してC:T=3:3条件にてz-scoreが高かった上位5つのパスウェイをTable. 3にまとめた。 DNAの修復やDNAの複製の安定化に関連するパスウェイや染色体分離、ATP生産に関連するパスウェイがC:T=3:3条件でアップレギュレートされていることが明らかになった。
 参考文献(16)より改変転載
参考文献(16)より改変転載4. 考察
本研究では、マルチオミクス解析により、CystineとTyr添加による培養トレンドと細胞内ストレスとの関係を明らかにした。Cystine単独の添加では、培養後半に生存率が減少、生産性が低下し、ERストレスがその原因となっていることがわかった。また、細胞内では、ERADの活性化およびアポトーシスの促進が確認された。CHOのFed-batch培養におけるCystine単独の添加は、細胞内での還元力を不足させ、細胞増殖・生産性に悪影響を及ぼすことが示唆された。
一方、Tyr単独の添加は、ERストレス・アポトーシスを抑制する効果があることが確認された。先行研究より、Tyr添加による生存率の維持は、オートファジー性細胞死の抑制効果によるものであるとされていたが(4)、P62/Sqstm1の発現量の結果から、Tyr枯渇条件での生存率の減少は、オートファジー誘導による細胞死ではないことが示唆された。
Tyrのアポトーシス抑制効果の検証として、Tyrから生合成されるコエンザイムQ10の添加実験を行ったところ、コエンザイムQ10が培養後半の生存率の維持に効果があることがわかった。つまり、コエンザイムQ10は細胞内ストレスやアポトーシスの抑制に効果があることが示唆された。ユビキノンは、主にミトコンドリアで活性があるとの報告がある一方で(36)、ユビキノンがゴルジ体やER膜に存在していることを示唆する報告もあり(37)、UBIAD1という酵素はミトコンドリア外にユビキノンを生産する酵素であることが報告されている(38)。今後、コエンザイムQ10のより詳細な効果の検証を行うために、ERでのストレス緩和のメカニズムの詳細を調べる必要があると考える。
一方、Tyrの単独添加は、生産性に大きな影響を与えなかった。これは、GSH利用の減少に対応するため、細胞代謝は、活性酸素の産生を抑えるために組換えタンパク質の産生を減少させたことに由来すると考えられる。この制御は、タンパク質のリン酸化等、この種のプロテオーム解析では検出されない他の制御機構に起因する可能性もあると考えられる。
次に、Cystineと同時にTyrを添加し、Cystineの量比を変えた実験を行ったところ、十分なCystineとTyrを添加することで、増殖能の向上・生存率の維持・生産性の向上を同時に達成できることが明らかとなった。ERストレスの抑制とGSH代謝の活性化やSOD1とSOD2による酸化ストレスへの適切な対処、ERADの減少およびTCAサイクルの活性化が増殖や生産性の向上に影響していることがわかった。また、CystineとTyrの十分量な添加は、PDIの発現をアップレギュレートすることがわかった。PDIA4はCHO細胞における抗体のジスルフィド結合形成を触媒することにより抗体生産に影響を与えることが知られており(39)、本研究で確認されたPDIA4の発現量の増加は、Komatsuらの結果と一致する。
また、C:T=1:3条件とC:T=3:3条件の増殖・生産性の差を明らかにするためにパスウェイ解析を行った。C:T=1:3条件に対してC:T=3:3条件にてz-scoreが高かった上位5つのパスウェイのうち3つのパスウェイは、DNAの修復やDNAの複製の安定化に関連するパスウェイであることが確認された。BER (Base Excision Repair) Pathwayは、内因性および外因性の変異原によって生じるDNA損傷および鎖切断の修復に関連するパスウェイであり、NER (Nucleotide Excision Repair, Enhanced Pathway) については、局所的な一本鎖DNA損傷を認識し、その損傷の除去に関連するパスウェイである。また、Cell Cycle Control of Chromosomal Replicationは、細胞分裂過程で起こるDNA複製を安定化させるパスウェイである。この結果から、C:T=3:3条件では、細胞はゲノムの安定性を高める機能を活性化させることで、培養後半の細胞にとって過酷な状況下においても、抗体生産を維持できる機能が働いていることが示唆された。また、C:T=3:3条件では、Kinetochore Metaphase Signaling Pathway とOxidative Phosphorylation が活性化されていることがわかった。Kinetochore Metaphase Signaling Pathwayは、キネトコアと微小管の相互作用を活性化し、細胞分裂の染色体分離を行うためのパスウェイであるため、細胞の増殖を促進する働きに関連していると考えられる。Oxidative Phosphorylationは、電子輸送系における電子の移動に由来するエネルギーを用いて細胞活動に欠かせないATPを生産するパスウェイである。ユビキノンは、ミトコンドリア膜内で電子をNADH-, FAD-dependent enzymesからRespiratory Complex IIIに輸送する役割がある(15)。このことから、Fig. 2 I, Jで示したコエンザイムQ10の添加による効果とOxidative Phosphorylationの活性化について、何らかの関係がある可能性が考えられるが、検証を行うためには、更なる研究が必要である。
5. 総括
CystineとTyrの添加が細胞ストレスやアポトーシスに与える影響をオミクス解析によって明らかすることができた。特に本研究では、CystineやTyr単独の要因による効果ではなく、2つのアミノ酸の交互作用を含む複雑な細胞メカニズムの詳細を明らかにした。また、培養条件間の比較により、CHO細胞による抗体生産には、CystineおよびTyrの供給比率と濃度が重要であることがわかった。
本研究のマルチオミクス解析で比較検討した各因子は、バイオプロセス中のストレスをモニタリングするための潜在的なマーカーになると考えられる。これらのマーカーを指標に培養プロセスの開発を行うことで、細胞ストレス軽減アプローチを通じてTyrに変わるコエンザイムQ10のような、代替添加物の可能性を特定することができると考える。
また、本研究で実施したマルチオミクス解析を活用した培養プロセス開発のアプローチは、Fed-Batch培養だけでなく、パーフュージョン培養等の様々な培養モードにも適用できると考えられ、応用範囲が広いと考えられる。今後、より複雑化することが予想される多様なモダリティのバイオ医薬品の生産プロセスを開発するにあたり、培養プロセス条件の変動に対する細胞内メカニズムの詳細を理解することが、バイオプロセスの生産性と堅牢な制御の向上にとって重要であると考える。
謝辞
本論文作成に際し、ご指導・ご鞭撻を賜りました東京農工大学 養王田 正文 教授に感謝申し上げます。
参考文献
- Walsh, Gary. “Biopharmaceutical benchmarks 2018.” Nature biotechnology vol. 36, 12 (2018): 1136-1145.doi:10.1038/nbt.4305
- Jeon, Min Kyoung et al. “Combinatorial engineering of ldh-a and bcl-2 for reducing lactate production and improving cell growth in dihydrofolate reductase-deficient Chinese hamster ovary cells.” Applied microbiology and biotechnology vol. 92, 4(2011): 779-90. doi:10.1007/s00253-011-3475-0
- Ali, Amr S et al. “Multi-Omics Study on the Impact of Cysteine Feed Level on Cell Viability and mAb Production in a CHO Bioprocess.” Biotechnology journal vol. 14, 4 (2019): e1800352. doi:10.1002/biot.201800352
- Tang, Hongping et al. “Insight into the roles of tyrosine on rCHO cell performance in fed-batch cultures.” Applied microbiology and biotechnology vol. 103, 16(2019): 6483-6494. doi:10.1007/s00253-019-09921-w
- Zeeshan, Hafiz Maher Ali et al. “Endoplasmic Reticulum Stress and Associated ROS.” International journal of molecular sciences vol. 17, 3 327. 2 Mar. 2016, doi:10.3390/ijms17030327
- Clement, G E, and T P Hartz. “Determination of the microscopic ionization constants of cysteine.” Journal of chemical education vol. 48, 6 (1971): 395-7. doi:10.1021/ed048p395
- Koppula, Pranavi et al. “Cystine transporter SLC7A11/xCT in cancer: ferroptosis, nutrient dependency, and cancer therapy.” Protein & cell vol. 12, 8(2021): 599-620.doi:10.1007/s13238-020-00789-5
- Chakravarthi, Seema, and Neil J Bulleid. “Glutathione is required to regulate the formation of native disulfide bonds within proteins entering the secretory pathway.” The Journal of biological chemistry vol. 279, 38 (2004:39872-9. doi:10.1074/jbc.M406912200
- Ribas, Vicent et al. “Glutathione and mitochondria.” Frontiers in pharmacology vol. 5 151. 1 Jul. 2014, doi:10.3389/fphar.2014.00151
- Ketterer, B et al. “The role of glutathione in detoxication.” Environmental health perspectives vol. 49(1983): 59-69. doi:10.1289/ehp.834959
- Chaudhari, Namrata et al. “A molecular web: endoplasmic reticulum stress, inflammation, and oxidative stress.” Frontiers in cellular neuroscience vol. 8 213. 29 Jul. 2014, doi:10.3389/fncel.2014.00213
- Ali, Amr S et al. “Multi-Omics Reveals Impact of Cysteine Feed Concentration and Resulting Redox Imbalance on Cellular Energy Metabolism and Specific Productivity in CHO Cell Bioprocessing.” Biotechnology journal vol. 15, 8(2020): e1900565. doi:10.1002/biot.201900565
- Yu, Marcella et al. “Understanding the intracellular effect of enhanced nutrient feeding toward high titer antibody production process.” Biotechnology and bioengineering vol. 108, 5(2011): 1078-88. doi:10.1002/bit.23031
- Feeney, Lauren et al. “Eliminating tyrosine sequence variants in CHO cell lines producing recombinant monoclonal antibodies.” Biotechnology and bioengineering vol. 110, 4(2013): 1087-97. doi:10.1002/bit.24759
- Meganathan, R. “Ubiquinone biosynthesis in microorganisms. ”FEMS microbiology letters vol. 203, 2 (2001): 131-9. doi:10.1111/j.1574-6968.2001.tb10831.x
- Shibafuji, Yusuke et al. “Cystine and tyrosine feed reduces oxidative and ER stress in CHO cells.” Biotechnology journal vol. 18, 7 (2023): e2200638. doi:10.1002/biot. 202200638
- Chen, Shifu et al. “fastp: an ultra-fast all-in-one FASTQ preprocessor.” Bioinformatics( Oxford, England) vol. 34, 17(2018): i884-i890. doi:10.1093/bioinformatics/bty560
- Dobin, Alexander et al. “STAR: ultrafast universal RNA-seq aligner.” Bioinformatics( Oxford, England) vol. 29, 1(2013): 15-21. doi:10.1093/bioinformatics/bts635
- Li, Bo, and Colin N Dewey. “RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome.” BMC bioinformatics vol. 12 323. 4 Aug. 2011, doi:10.1186/1471-2105-12-323
- Robinson, Mark D et al. “edgeR: a Bioconductor package for differential expression analysis of digital gene expression data.” Bioinformatics(Oxford, England) vol. 26, 1(2010): 139-40. doi:10.1093/bioinformatics/btp616
- Gessulat, Siegfried et al. “Prosit: proteome-wide prediction of peptide tandem mass spectra by deep learning.” Nature methods vol. 16, 6(2019): 509-518. doi:10.1038/s41592-019-0426-7
- Yoshida, H et al. “Identification of the cis-acting endoplasmic reticulum stress response element responsible for transcriptional induction of mammalian glucose-regulated proteins. Involvement of basic leucine zipper transcription factors.” The Journal of biological chemistry vol. 273, 50 (1998): 33741-9. doi:10.1074/jbc.273.50.33741
- Hetz, Claudio. “The unfolded protein response: controlling cell fate decisions under ER stress and beyond.” Nature reviews. Molecular cell biology vol. 13, 2 89-102. 18 Jan. 2012, doi:10.1038/nrm3270
- Kilberg, M S et al. “Nutritional control of gene expression: how mammalian cells respond to amino acid limitation.” Annual review of nutrition vol. 25 (2005): 59-85. doi:10.1146/annurev.nutr.24.012003.132145
- Hetz, Claudio et al. “Targeting the unfolded protein response in disease.” Nature reviews. Drug discovery vol.12, 9(2013): 703-19. doi:10.1038/nrd3976
- Liang, Jing et al. “Characterization of erasin(UBXD2): a new ER protein that promotes ER-associated protein degradation.” Journal of cell science vol. 119,Pt 19(2006):4011-24. doi:10.1242/jcs.03163
- Sun, Shengyi et al. “Sel1L is indispensable for mammalian endoplasmic reticulum-associated degradation, endoplasmic reticulum homeostasis, and survival.” Proceedings of the National Academy of Sciences of the United States of America vol. 111, 5 (2014): E582-91. doi:10.1073/pnas.1318114111
- Liu, Lintao et al. “GADD34 Facilitates Cell Death Resulting from Proteasome Inhibition.” Anticancer research vol. 35, 10(2015): 5317-24.
- Bjørkøy, Geir et al. “Monitoring autophagic degradation of p62/SQSTM1.” Methods in enzymology vol. 452(2009): 181-97. doi:10.1016/S0076-6879(08)03612-4
- Fukui, Masayuki, and Bao Ting Zhu. “Mitochondrial superoxide dismutase SOD2, but not cytosolic SOD1, plays a critical role in protection against glutamate-induced oxidative stress and cell death in HT22 neuronal cells.” Free radical biology & medicine vol. 48, 6(2010): 821-30. doi:10.1016/j.freeradbiomed.2009.12.024
- Zelko, Igor N et al. “Superoxide dismutase multigene family: a comparison of the CuZn-SOD(SOD1), Mn-SOD(SOD2), and EC-SOD(SOD3) gene structures, evolution, and expression.” Free radical biology & medicine vol. 33, 3(2002): 337-49. doi:10.1016/s0891-5849(02)00905-x
- Paolicchi, Aldo et al. “Glutathione catabolism as a signaling mechanism.” Biochemical pharmacology vol. 64, 5-6(2002): 1027-35. doi:10.1016/s0006-2952(02)01173-5
- Xiang, Zhidan et al. “Mice lacking three Loci encoding 14 glutathione transferase genes: a novel tool for assigning function to the GSTP, GSTM, and GSTT families.” Drug metabolism and disposition: the biological fate of chemicals vol. 42, 6(2014): 1074-83. doi:10.1124/dmd.113.056481
- Rutter, Jared et al. “Succinate dehydrogenase - Assembly, regulation and role in human disease.” Mitochondrion vol. 10, 4(2010): 393-401. doi:10.1016/j.mito.2010.03.001
- Lushchak, Oleh V et al. “Aconitase post-translational modification as a key in linkage between Krebs cycle, iron homeostasis, redox signaling, and metabolism of reactive oxygen species.” Redox report : communications in free radical research vol. 19, 1(2014): 8-15. doi:10.1179/1351000213Y.0000000073
- Wang, Ying, and Siegfried Hekimi. “Understanding Ubiquinone.” Trends in cell biology vol. 26, 5(2016): 367-378. doi:10.1016/j.tcb.2015.12.007
- Kalén, A et al. “Nonaprenyl-4-hydroxybenzoate transferase, an enzyme involved in ubiquinone biosynthesis, in the endoplasmic reticulum-Golgi system of rat liver.” The Journal of biological chemistry vol. 265, 2(1990): 1158-64.
- Mugoni, Vera et al. “Ubiad1 is an antioxidant enzyme that regulates eNOS activity by CoQ10 synthesis.” Cell vol. 152, 3(2013): 504-18. doi:10.1016/j.cell.2013.01.013
- Komatsu, Kei et al. “Effect of the disulfide isomerase PDIa4 on the antibody production of Chinese hamster ovary cells.” Journal of bioscience and bioengineering vol. 130, 6(2020): 637-643. doi:10.1016/j.jbiosc.2020.08.001